#install.packages("palmerpenguins")
library(palmerpenguins)
data("penguins")
d <- penguins penguins-vis-bodymass2
vis
Aufgabe
Im Datensatz palmerpenguins: Ist der Zusammenhang zwischen Körpergewicht und Schnabelhöhe (bill depth) (vgl. Schemazeichnung hier) größer, wenn man den Zusammenhang getrennt für jede Spezies betrachtet?
Beantworten Sie diese Frage mit Hilfe einer Visualisierung!
Sie können den Datensatz so beziehen:
Oder so:
d <- read.csv("https://vincentarelbundock.github.io/Rdatasets/csv/palmerpenguins/penguins.csv")Ein Codebook finden Sie hier.
Hinweise:
- Orientieren Sie sich im Übrigen an den allgemeinen Hinweisen des Datenwerks.
- Nutzen Sie das R-Paket
ggpubrzur Visualisierung. Dort finden Sie einen Befehl namensggscatter(datensatz, x-variable, y_variable, facet_by), mit dem Sie Streudiagramme aufgeteilt nach (“facettiert nach”) einer (nominal skalierten) Gruppierungsvariablen erstellen können.
Lösung
library(tidyverse)── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ dplyr 1.1.4 ✔ readr 2.1.5
✔ forcats 1.0.0 ✔ stringr 1.5.1
✔ ggplot2 3.5.0 ✔ tibble 3.2.1
✔ lubridate 1.9.3 ✔ tidyr 1.3.1
✔ purrr 1.0.2
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errorslibrary(ggpubr)d <- read.csv("https://vincentarelbundock.github.io/Rdatasets/csv/palmerpenguins/penguins.csv")d |>
ggscatter(x = "bill_depth_mm", y = "body_mass_g", facet.by = "species")Warning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).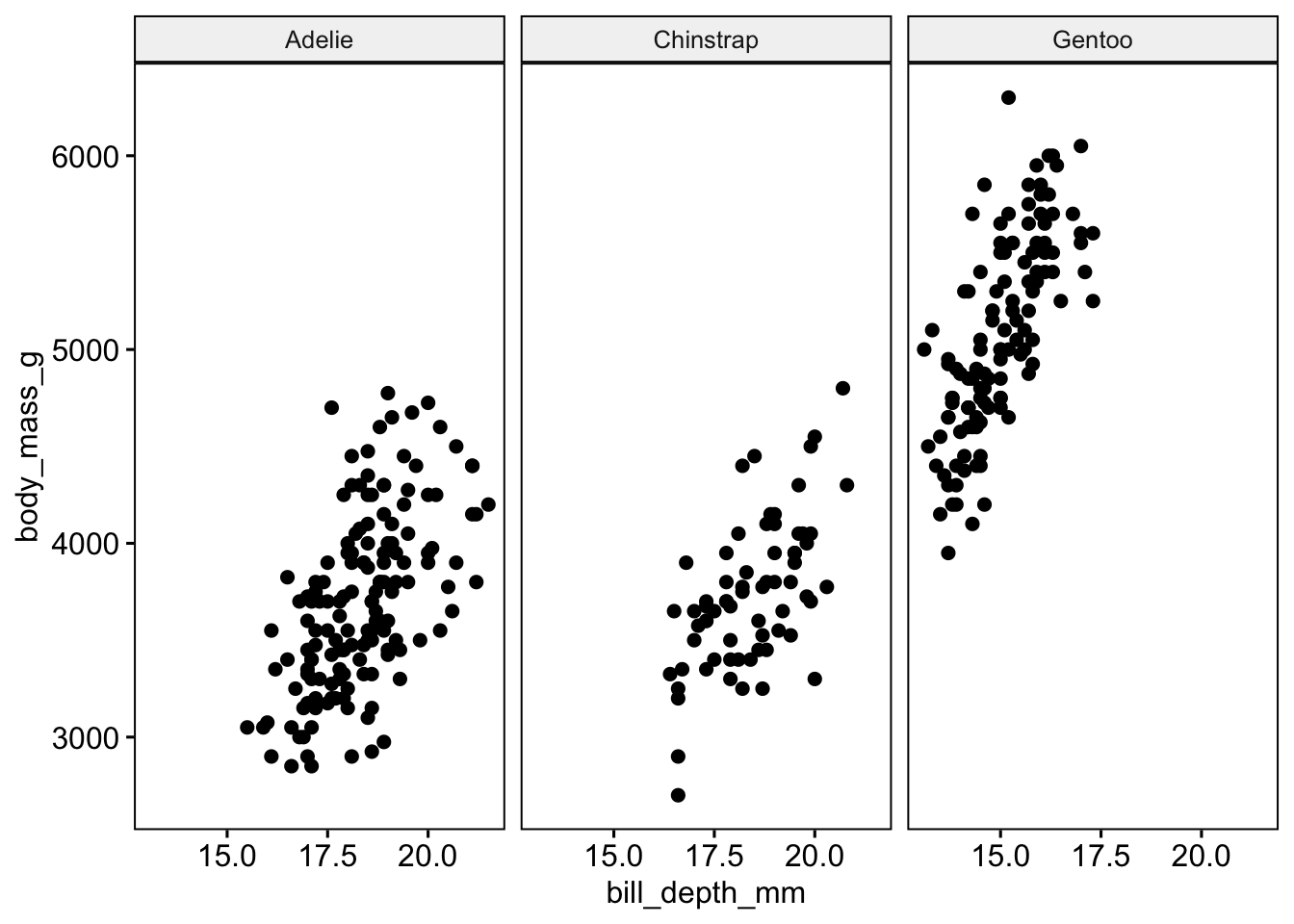
Und jetzt erstellen wir das Streudiagramm ohne Aufteilung in die Gruppen von species:
ggscatter(d, x = "bill_depth_mm", y = "body_mass_g")Warning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).
Wie man sieht, tritt der Zusammenhang klarer hervor, wenn man die Daten in die von species definierten Gruppen aufteilt.