library(tidymodels)
data(penguins, package = "palmerpenguins")tidymodels-poly02
R
statlearning
tidymodels
num
Aufgabe
Fitten Sie ein Polynomial-Modell für folgende Modellgleichung:
body_mass_g ~ bill_length_mm.
Gesucht ist der RMSE im Test-Set (optimal hinsichtlich minimalem Prognosefehler).
Hinweise:
- Datensatz
penguins(palmerpenguins) - Verwenden Sie Tidymodels
- Fitten Sie Polynome des Grades 1 bis 10.
- Definieren Sie die Polynomegrade als Tuningparameter.
- Entfernen Sie fehlende Werte in den Prädiktoren.
- Wie immer gilt: Verwenden Sie die Standardeinstellungen der Funktionen, soweit nicht anders angegeben.
Lösung
Setup:
Datenaufteilung:
d_split <- initial_split(penguins)
d_train <- training(d_split)
d_test <- testing(d_split)Rezept:
rec1 <-
recipe(body_mass_g ~ bill_length_mm, data = penguins) %>%
step_naomit(all_predictors()) %>%
step_poly(all_predictors(), degree = tune()) %>%
update_role(contains("_poly_"), new_role = "predictor")Warning: No columns were selected in `update_role()`.Check:
d_baked <- bake(prep(rec1), new_data = NULL)Rezepte mit Tuningparametern kann man nicht preppen/backen.
Workflow:
wf1 <-
workflow() %>%
add_model(linear_reg()) %>%
add_recipe(rec1)Tuning:
set.seed(42)
tune1 <-
tune_grid(
wf1,
resamples = vfold_cv(data = penguins),
metrics = metric_set(rmse),
grid = grid_regular(degree(range = c(1, 10)),
levels = 10),
control = control_grid(save_workflow = TRUE)
)autoplot(tune1)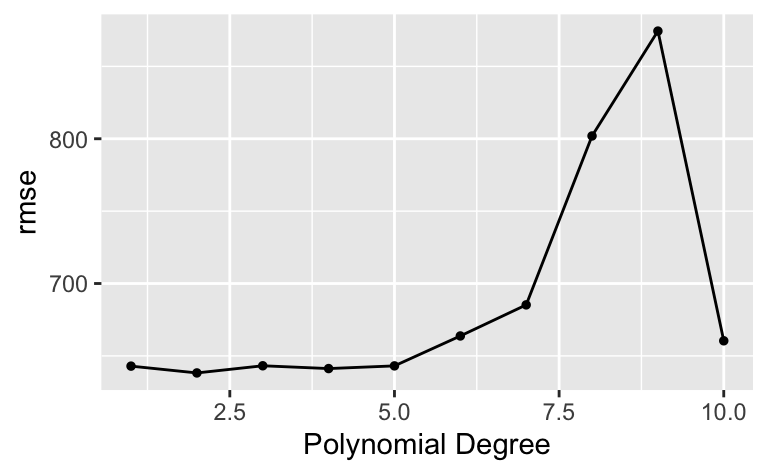
show_best(tune1)# A tibble: 5 × 7
degree .metric .estimator mean n std_err .config
<dbl> <chr> <chr> <dbl> <int> <dbl> <chr>
1 2 rmse standard 638. 10 22.7 Preprocessor02_Model1
2 4 rmse standard 641. 10 23.7 Preprocessor04_Model1
3 1 rmse standard 643. 10 21.8 Preprocessor01_Model1
4 5 rmse standard 643. 10 23.5 Preprocessor05_Model1
5 3 rmse standard 643. 10 24.2 Preprocessor03_Model1Finalisieren:
best1 <- fit_best(tune1)
best1══ Workflow [trained] ══════════════════════════════════════════════════════════
Preprocessor: Recipe
Model: linear_reg()
── Preprocessor ────────────────────────────────────────────────────────────────
2 Recipe Steps
• step_naomit()
• step_poly()
── Model ───────────────────────────────────────────────────────────────────────
Call:
stats::lm(formula = ..y ~ ., data = data)
Coefficients:
(Intercept) bill_length_mm_poly_1 bill_length_mm_poly_2
4202 8813 -1708 Predicten:
final1 <- last_fit(best1, d_split)
collect_metrics(final1)# A tibble: 2 × 4
.metric .estimator .estimate .config
<chr> <chr> <dbl> <chr>
1 rmse standard 666. Preprocessor1_Model1
2 rsq standard 0.287 Preprocessor1_Model1Oder so:
sol <-
predict(best1, new_data = d_test) %>%
bind_cols(d_test) %>%
rmse(truth = body_mass_g, estimate = .pred) %>%
pull(.estimate) %>%
pluck(1)
sol[1] 657.5297Die Antwort lautet: 657.5296534.
Categories:
- R
- statlearning
- tidymodels
- num