library(tidyverse)
library(easystats)
library(rstanarm)
data("penguins", package = "palmerpenguins")penguins-stan-04a
Aufgabe
Wir untersuchen Einflussfaktoren bzw. Prädiktoren auf das Körpergewicht von Pinguinen. In dieser Aufgabe untersuchen wir den Zusammenhang von Schnabellänge (als UV) und Körpergewicht (als AV).
Aufgabe:
Wie groß ist die Wahrscheinlichkeit, dass der Effekt vorhanden ist (also größer als Null ist), die “Effektwahrscheinlichkeit”? Geben Sie die Wahrscheinlichkeit an.
Hinweise:
- Nutzen Sie den Datensatz zu den Palmer Penguins.
- Verwenden Sie Methoden der Bayes-Statistik und die Software Stan.
- Sie können den Datensatz z.B. hier beziehen oder über das R-Paket
palmerpenguins. - Weitere Hinweise
Setup:
Es wird in dieser Aufgabe vorausgesetzt, dass Sie den Datensatz selbständig importieren können. Tipp: Kurzes Googeln hilft ggf., den Datensatz zu finden.
Alternativ könnten Sie den Datensatz als CSV-Datei importieren:
d_path <- "https://vincentarelbundock.github.io/Rdatasets/csv/palmerpenguins/penguins.csv"
penguins <- data_read(d_path)Ein Blick in die Daten zur Kontrolle, ob das Importieren richtig funktioniert hat:
glimpse(penguins)Rows: 344
Columns: 8
$ species <fct> Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, Adel…
$ island <fct> Torgersen, Torgersen, Torgersen, Torgersen, Torgerse…
$ bill_length_mm <dbl> 39.1, 39.5, 40.3, NA, 36.7, 39.3, 38.9, 39.2, 34.1, …
$ bill_depth_mm <dbl> 18.7, 17.4, 18.0, NA, 19.3, 20.6, 17.8, 19.6, 18.1, …
$ flipper_length_mm <int> 181, 186, 195, NA, 193, 190, 181, 195, 193, 190, 186…
$ body_mass_g <int> 3750, 3800, 3250, NA, 3450, 3650, 3625, 4675, 3475, …
$ sex <fct> male, female, female, NA, female, male, female, male…
$ year <int> 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007…Vertrauen ist gut, aber - was Golems betrifft - ist Kontrolle eindeutig besser ;-)
m1 <- stan_glm(body_mass_g ~ bill_length_mm, # Regressionsgleichung
data = penguins, # Daten
seed = 42, # Repro.
refresh = 0) # nicht so viel Outputparameters(m1)| Parameter | Median | CI | CI_low | CI_high | pd | Rhat | ESS | Prior_Distribution | Prior_Location | Prior_Scale |
|---|---|---|---|---|---|---|---|---|---|---|
| (Intercept) | 362.87728 | 0.95 | -192.71794 | 899.6088 | 0.906 | 0.9997420 | 3918.566 | normal | 4201.754 | 2004.8863 |
| bill_length_mm | 87.45409 | 0.95 | 75.22585 | 100.1245 | 1.000 | 0.9997733 | 3914.346 | normal | 0.000 | 367.2233 |
ETI:
eti(m1) |> plot()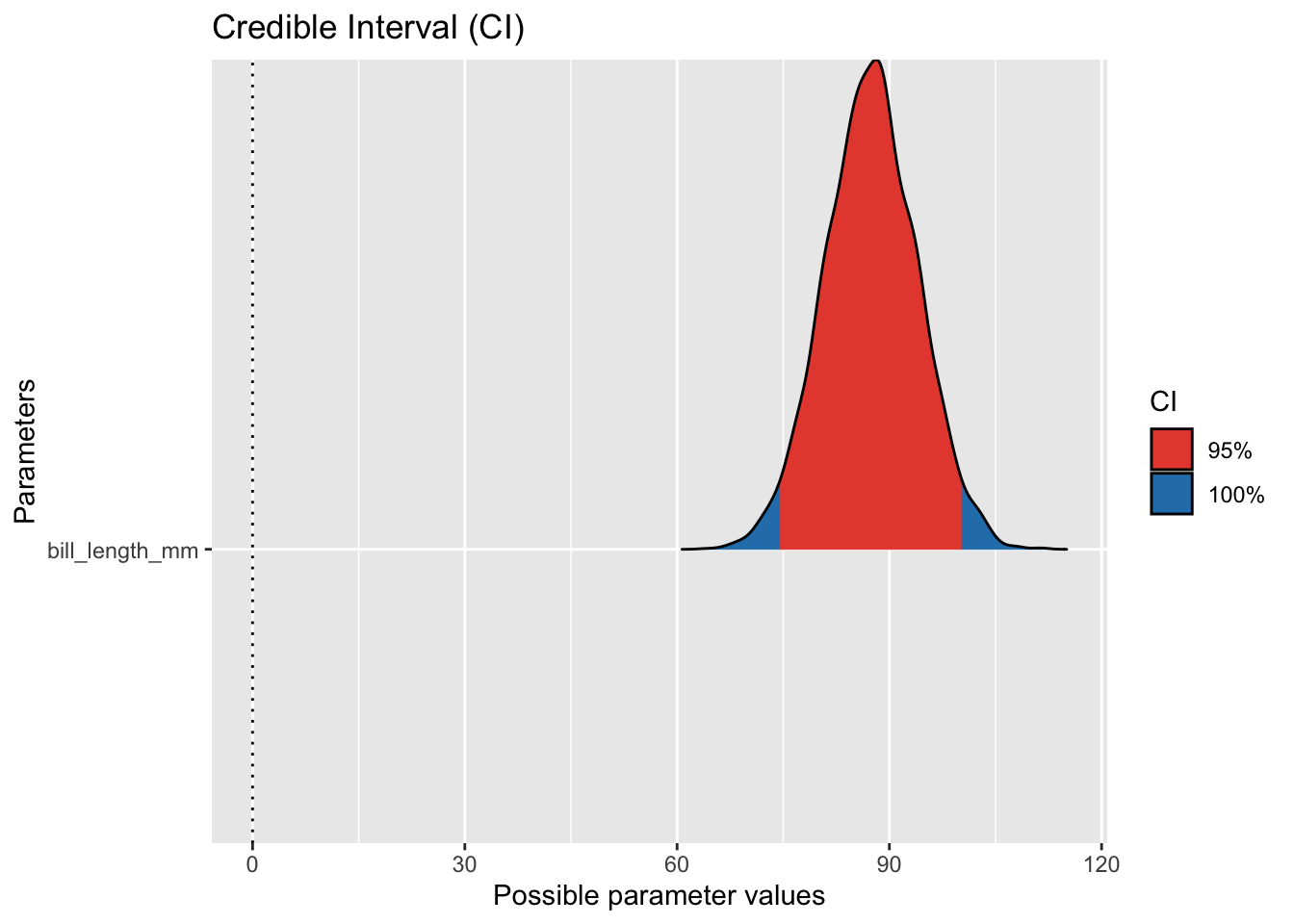
Lösung
Man sieht im Diagramm direkt, dass 100% des Schätzbereichs rechts von der Null ist. Daher ist die Effektwahrscheinlichkeit 100%.
Alternativ bekommt man die Statistik auch mit parameters(), wie in der Tabelle oben in der Spalte pd ersichtlich.
Mit pd() kann man sich die Effektwahrscheinlichkeit (“probability of direction”) ausgeben lassen:
pd(m1)| Parameter | pd | Effects | Component |
|---|---|---|---|
| (Intercept) | 0.906 | fixed | conditional |
| bill_length_mm | 1.000 | fixed | conditional |
Mehr Informationen zu dieser Statistik findet sich hier oder hier.
Die Lösung lautet also 1.
Categories:
- bayes
- regression
- exam-22